2019.04.09
YCAM InterLab Camp vol.3「パーソナル・バイオテクノロジー」レポート#2 — Bento Labのストーリー、DNAの「読み」「書き」のストーリー
[編注:レポート#1はこちら]
集中ワークショップ「パーソナル・バイオテクノロジー」の2日目の集合時間は、午前9時半。会場に入るとすぐに足を向けたのは、昨夜から稼働しているPCの前だ。10時間以上、しっかり動き続けてくれている。
ビューアーのタイルの色は、昨日より深緑色が増えている(前回記事参照)。タイルひとつずつは、シーケンサー「MinION」のナノポアを表していて「明るい緑色はナノポアが良好」という話だったけれど、こんなに緑が濃くなっていて大丈夫なのだろうか? 心配顔をしていたら、「深緑色はシーケンシングはできる状態でもDNAが入っていないだけ。時間経過でこうなっていくんですよ」と教えてもらった。スタッフは画面を動かして、別のデータ画面も見せてくれた。「けっこうロングリードも取れていますよ」。それなら安心! 見えないものに相対しているわれわれの一喜一憂は、まずはスタッフの一言で決まる。
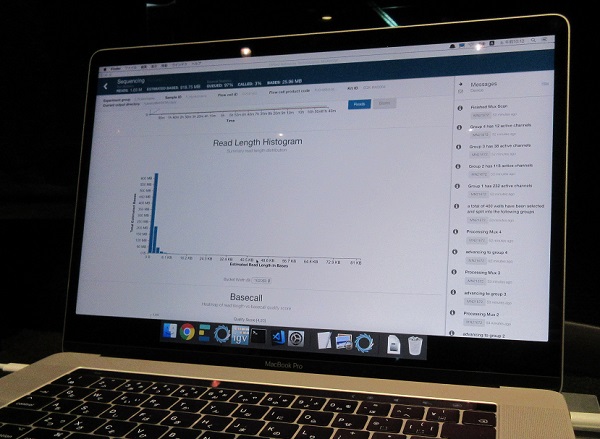
読まれた塩基対の長さのグラフ。MBやKBという単位が見えるけれど、この「B」は「バイト」ではない。ベース・ペア(base pairs:Bp)の略らしい
DNA配列の解析―シーケンシングしたばかりのデータを自分たちで解析してみる
2日目のプログラム1本目は、ライフサイエンス統合データベースセンター(DBCLS)の特任助教片山俊明氏による「バイオインフォマティクス」。ゲノム情報など生命科学に関わる情報を統合的に利用するためのソフトウェアを研究開発している氏から、昨日来読み取っているDNAシーケンスの解析方法を学ぶ。
まず、ゲノム弁当の発案者のひとりでもある片山氏から、ゲノム弁当の企画概要と「ゲノム解読」の意味が解説された。次に、氏が愛してやまない不思議生物、クマムシの生態と「クマムシゲノムプロジェクト」の紹介。国内外の各地で採取されたクマムシのゲノムを研究者が連携して解読することで系統関係を明らかにしていくというプロジェクトの目的や活動から、ゲノムデータべースの活用法などを何となくイメージする。
そのうえでいよいよ、MinIONで読んだデータを見ていくことになった。以下は、その手順である。
ゲノムのマッピング
・リードを参照配列にマッピング
読んだデータ(リード)は塩基配列の断片であるため、最初に「参照配列」を準備してマッピング(アライメント)をする方法を使う。それに使うのが、マッピングツールだ。「ロングリードに対応したツールはまだ少なめ」だそう。今回は、MinIONによりロングリードが読めているので、「BWA」というツールを使用する。
|あらかじめの下準備|
公開されているデータベースなどから参照配列ファイルを準備する。今回、このファイルはすでに準備されていた。
|FASTQファイルを探す|
手元にあるMinIONの生データから、「FASTQ」という形式に変換されたデータを探して、それを使う。FASTQとは、リード(配列断片)の標準的なファイル形式で、MinIONに付属の「MinKNOWソフトウェア」がMinION特有ファイル形式「fast5」から自動変換してくれる。以前はツールを使って自分で変換する必要があったから、だいぶ便利になったということだ。
|BWAを用いてマッピング|
ロングリードのマッピングにも対応したbwaコマンドを用いてマッピング。できるファイルは「SAM」ファイルとなる。
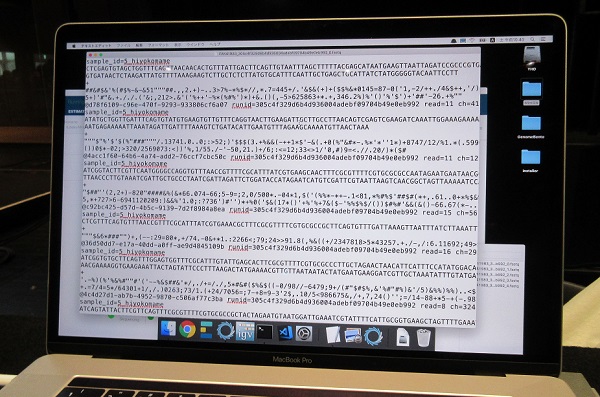
デスクトップより探し出されたひよこ豆DNAのFASTQファイル。これを探すだけでも初心者はなかなかたいへん

片山先生の解説スライドより。上の写真の画面のデータと見合わせると、@マーク付きの配列IDの次の行から、「2.塩基配列」データが取れていることがわかる
・IGVを使ってマッピング結果を見てみる
できたSAMファイルを可視化するツールが「IGV」。IGVは、リファレンスゲノムおよびマッピングされたリードを表示することができ、ヒトゲノムのように巨大なゲノムにも対応している。IGVは、起動して参照配列を読み込んでおく。
|SAMファイルのソートとインデックス作成|
SAMファイルは、できたままだとマッピングされたリードがゲノム座標順に並んでいないので、IGVで見るには効率が悪い。このため、通常はソートしてインデックス作成したものを利用する。ここではIGVに内蔵されている「igvtools」というものを使う。
|マッピング結果を読み込む|
マッピングの状況を見ながら結果が出ていそうなところでズームしていくと、マッピングされたリードが出てくる。

こうした解析の流れはまとめるとこのようになる。マッピングと可視化をうまくやるためには、データのファイル形式を何度も変換して対応させていく必要がある
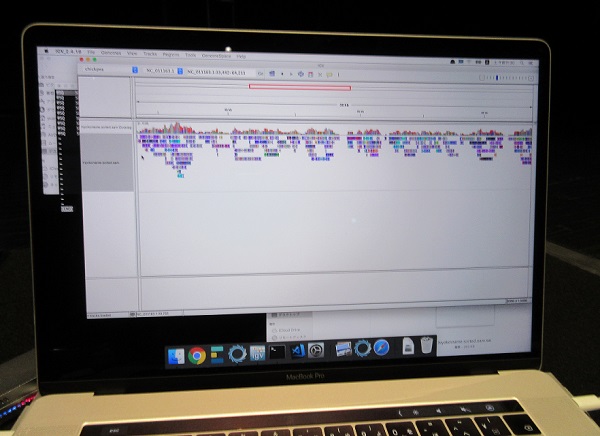
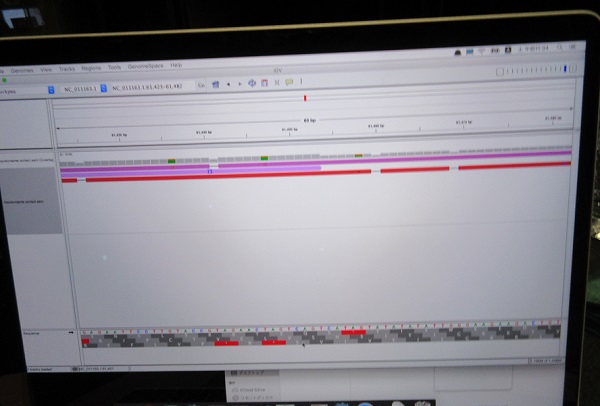
結果として、このようなマッピングと可視化ができた。ひよこ豆は「運よく」長いリードが取れていて、ひよこ豆であったことが判明!
以上の作業を、グループごとに画面をのぞき込み、スタッフの助けも借りながら進めていった。そのうちに、となりの席の生物学系の学生は、「ちょっと…わかりにくいですね…」とつぶやくのである。ええ、私も同様です。向かいの席の工学系の院生は、「ちょっと…もうちょっと便利なものがないのかな…」とつぶやく。ええ、ここは工学的な一気通貫ツールがあってよい気がしてなりませんね。
そのとき壇上の片山氏が、「ここ、ここはもっと使いやすいツールの登場が待たれるところなんです!」とおっしゃった。私などは、バイオインフォマティックスに興味を持つ各分野のテクニックと開発力を持った人々がどうか増えますように、と望むばかりである。
ゲノムのデータベース
次に、「未来のゲノム弁当」に食材を追加する方法、つまり食材にしたいもののゲノム解読状況を調べ、ない場合には新たに解析データを登録する方法が紹介された。ゲノムデータベースとしては、現在以下のようなものがある。
・解読されたゲノム配列を網羅的に収集したデータベース NCBI Genome
・ヒトゲノムの主要なデータベース UCSC Genome Browser
・主要な生物種を広くカバーするゲノムデータベース Ensembl, TogoGenome
・ゲノム解読の進行している生物種を網羅したデータベース JGI GOLD
最新の解読情報は、論文検索でわかることも多いそうだ。お話を聞いていると、自分が採取したデータを世の中のデータベースと組み合わせて、巨大データベースの一角に個人も参画できる可能性があるようだ。そこに、バイオインフォマティックスの醍醐味もあるように思われてきた。

採れたてデータをほぼリアルタイムで解析しながらプレゼン、ご指導してくださった片山先生。先生はどれほどの事前準備をしたものだろうか、昨夜は眠れたのだろうか、ありがとうございます(撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])
トークのダイジェスト1 ― フィリップ・ボーイング氏(Bento Bioworks)
ますます味わい深く感じられるようになったゲノム弁当のランチの後、寝落ち寸前となったわれわれの目を覚まさせてくれたのは、個人サイエンティストのセバスチャン・コシオバ氏のトークである。その内容を紹介する前に、「Bento Bioworks」共同設立者のフィリップ・ボーイング氏のトークも紹介しておきたい。ボーイング氏のトークは、1日目の実験中、それもBento LabのPCRが絶賛稼働中に開発者ご当人のお話を聞くという、イキなはからいのプログラム構成で行われた。
Bento Labのアイデアが生まれるまで
ロンドンの大学で計算機科学を専攻する学生だったボーイング氏は、合成生物学パーツであるところの「バイオブリック」が登場した頃から、生物のプログラミング可能性に本格的に興味を持つようになった。標準化されたバイオブリックを活用するコンペティション「iGEM」(国際合成生物学大会)の活動に関わるようになり、大学の4年間はiGEMの活動に没頭した。
大学時代は大学のラボが使えたのだが、卒業すると活動が難しくなってしまう。そこでボーイング氏は、iGEM活動の仲間、べサン・ウルフェンデンさん(Bento Bioworksの共同創業者)とともに、イーストロンドンの「HackerSpace」で、DIY BIOをやっていくことになった。とはいえ、HackerSpaceはメイカースペースだから、バイオラボではない。まずはアイデア出しや将来の活動の夢などを話し合うくらいだったのだけれども、そのうちに自分たちでもバイオブリックを作って「標準生物学的パーツレジストリ」[注:バイオブリックの登録先。いわばバイオブリックカタログ]に登録しよう、ということになった。
そこでボーイング氏らは、ゲル電気泳動ユニット、細胞培養器、インキュベーターといった実験機材を手作りしながらDNAを取り出せる環境を整えていき、バイオブリックを登録をすることに成功した。その登録は、大学以外のDIYバイオを指向するメイカースペースからは“初”のことだったそうだ。

コイル、スイッチボックス、ライトをつけて手作りしたインキュベーター。Bento Labの最初期原型のようなもの。「大学のラボならこうした機材は立派なものがそろっているのがあたり前。でもHackerSpaceには何もないから作ったんです」
このプロジェクトを博物館のギャラリーで展示したり、各地のメイカースペースやファブラボで話したりしていたら、ボーイング氏は質問されるようになっていった。その質問はだいたい同じなFAQで、「自分たちもDIY BIOをやりたいけど、どうやったら始められるの?」なのだった。どんな機材が必要なの、それはどこで調達できるの、どんなプロジェクトから始めればいいの、といったところ。
その一方で、当時はオープンソースの設計図から組み立て可能なPCR「OpenPCR」が、メイカースペースやファブラボで作られるようになっていた。しかしこれは機械工学や電気工学の知識がかなり必要で、そう手軽ではない。また当時、メイカースペースで話題になっていたトピックには、「ブリーフケースラボ」のコンセプトがあった。それは、「ブリーフケースの中に様々な工具やマシンを詰め込んで移動できるモバイルなラボ」だ。
ボーイング&べサン両氏の頭の中には、「ブリーフケースの中にバイオラボを入れよう!」がひらめいた。
Bento Labのはじまり
2012年にRaspberryPiが発売されたことから、アイデアは現実味を帯びていく。両氏はまず、スケッチを書き始めた。それは例えば、森の中にテントを張ってラボを作り実験、実験を終えたらテントをたたんで撤収する、というようなもの。
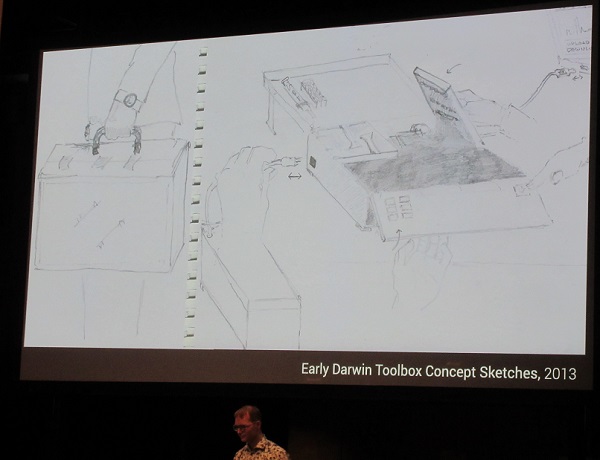

スケッチとコンセプト。「このDarwin Toolbox Concept(2013)は、まだアイデアです。でも、このアイデア段階のものを各地のメイカースペースで見せて話をしたり、ローマのMaker Faireに出展したりました」
そうするうちにコンセプトは練られていき、A4サイズで名称も「Bento Lab」になっていく。2014年にはモデル機(といってもモックアップみたいなもの)ができたけれど、まだきちんと稼働はしない。
奨学金を得て日本に9カ月間留学したボーイング氏は、メディアアートとバイオロジーにまたがって活動するアーティストグループ「BCL」に参加、屋台ラボ「Oubiopo」を作ったり、子ども向けのバイオイベント「バイオ祭」を行ったりしていた。Bento Labはウェブサイトに情報を載せていたのだが、その間も「これはどこで買えるの?」という問い合わせが続々と来ていた。
深圳のMaker Faireに招待されて参加をした時も、まだアイデアだけ。なのにみんなが「どこで買えるの?」と聞いてくる。東京でスタートアップのコンペティション「SLUSH Asia」に出してみたところ、Bento Labはファイナリストの5作に残ることができた。いよいよ、「これはホンモノの会社を作ってホンモノを作っていくべきなのではないか」と考えるようになったボーイング&べサン両氏。ついにKickstarterの準備を始めることにした。「私たちは、これがよいアイデアであるだけでなく、本当に役に立つものだということを示したかったんです。実際に作って、それを証明したいと思いました」
Kickstarterキャンペーンと生産の準備
Bento Labは、ソフトウェアが動くようになった。PCRは温度の微妙な管理が難しかったけれど、なんとかクリア。まずはその程度の機能で、20台の試作機を製作。オープンにベータテスト(2015-2016年)を呼びかけた。
それから20台の使用事例をベースに、Kickstarterキャンペーン開始(2016年3月~)。と、目標金額の4万ポンドを大幅に上回る約15万ポンドの出資金を得ることができた。「私たちは資金を得ました。しかし、今度は作らなければならないんです! 実機の製造にこぎつけるまで、そこから2年以上がかかってしまいました」
課題は多かった。ベータテストのフィードバックからの設計変更、もはやハンドメイドでは不可能なのでサプライヤー探し。そもそもの会社の創業や運営、コストの問題、エンジニアのマネジメント……すべてを知らないのでひとつひとつを学び、実現に結びつけていくしかなかった。最初にできてきたBento Labは、届いてすぐに壊れてしまったので遠隔地での製造は無理だと判断、国内での製造に切り替えて、ようやく実機が見えてきた。
「2018年末に、ようやく出荷ができるようになりました。しかし、私たちの目標はハードウェアだけではないのですね。インストラクターがいなくても、Bento Labを入手した人が自分でプロジェクトを展開できるようになること、本当のDIYバイオが私たちの目標なんです。なので、Bento Labはスターターキットもセットにして販売していきます」
スターターキットができたのは、つい先頃の2月末だそうだ。「これまで私たちがたくさんの人たちから聞かれてきたDIYバイオについての質問に、このBento Labとスターターキットで答えていきたい」と、ボーイング氏は語った。



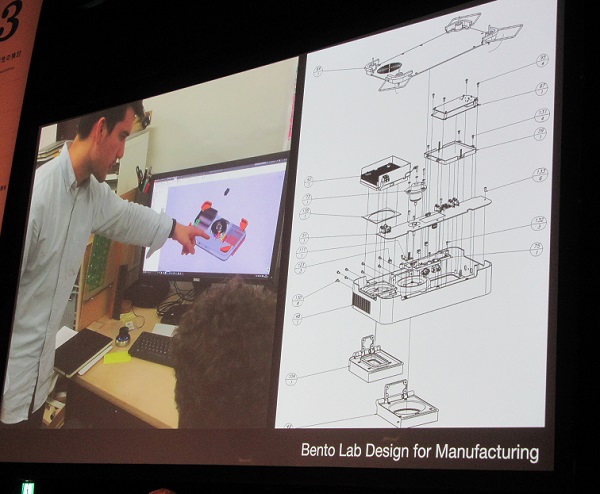

上からモックアップ、デモ機、生産のための設計変更、社内生産の様子。Bento LabBento Labはこのように進化してきた。「現在は、Bento Bioworks社内で週に20台の製造体制」

できたてのスターターキット

製造されたばかりのBento Lab2台を手持ちしてロンドンよりワークショップに駆けつけたボーイング氏。「入国審査はどうでしたか?」の会場からの質問には、「どう説明しようとすごく考えてきたのだけど、実験の機械だと言ったらすんなりでしたよ」とのこと(撮影:伊藤隆之(YCAM)、写真提供:山口情報芸術センター[YCAM])
トークのダイジェスト2 ― セバスチャン・コシオバ氏(Binomica Labs)
セバスチャン・コシオバ氏は、合成生物学を手がける個人サイエンティストだ。専門的に学んだことはなく、独学でここまで来たという。主宰しているBinomica Labsはボランティアグループによって運営されていて、オープンな研究とオープンなツールやリソースの提供を信条としている。
そのラボがまた独特で、それはお母さんが住むニューヨークのアパートメントの一角(第三寝室)を改装して研究所としている。米国のバイオラボには「バイオセーフティレベル」という取り扱いレベルの規制があるが、Binomica Labsは「レベル1」[注:高校の実験室程度]。住居を兼ねても危険はなく、そのような設備でもハイレベルの研究ができることを、コシオバ氏自らが証明しているということになる。
またコシオバ氏は、物心ついたときから植物に関心を持ち、プランターやポットで植物を育成し続けている。この育成も住居内で行われていて、そうした植物が「実験のワークベンチであり、私の実験のシンボル」なのだそうだ。そんな氏は、Binomica Labsが手がけてきた「読み」のプロジェクトを2つ、「書き」のプロジェクトを2つ紹介してくれた。


高層ビルに囲まれた住居内のラボ。氏が初めて交配したこの花は、毎日違う色の花が咲く。「間違えて鉢を壊してしまったことから、偶然にできました」
「読み」のプロジェクト1―オキサリス(カタバミ)
「オキサリス」は雑草で、和名はカタバミ。ある日、レモンツリーを育てていたポットの中に芽吹いたこの雑草をもっと詳しく知ろうと調べてみたけれど、あまりにありふれているために研究対象になっていない。世界中に分布していて、色も大きさのバリエーションも多様。「コスモポリタンな生命体だ」と感じた氏は、生物学的に形態学的にこの植物がどんな変化をしているのかを見極めるため、世界中のカタバミのゲノム解読プロジェクトを始めた。
このプロジェクトには、サイドストーリーも芽生えることになった。氏は、植物学や組織培養方法を市民に教えるワークショップも行っているのだが、そこに当時10歳のビアンカさん(現在12歳)が参加した。彼女が子どもサイエンスフェアに参加するというので、氏は「理科の実験なんて昔から同じで退屈だ。僕と一緒にカタバミを研究してみない?」と持ちかけた。「カタバミを増やしたいんだけどうまく増えないんだよ」と言ったら彼女、「いったい何を食べて生きているの? それを調べてみたい」となり、次のような実験をしていった。
・肥料成分5つを混合、濃度を変えて、100本のカタバミの成育状況を調べた
・実験ノートを取りながら、細かく記録。タイムラプスで毎日ツイッターに公開した
・生育したカタバミの成分を詳細に分析した
・統計方法を教えてあげたら報告が届き、サイエンスフェアでは1等賞!
つまり、10歳の女の子がカタバミの必須栄養素量を明らかにしたのだ(もちろん世界初!)。彼女の実験は、カタバミのゲノム解読にも貢献した。植物のゲノム解読は組織培養された純粋な植物細胞がないとできない(土や細菌が付着しているとうまくいかない)。彼女の実験はカタバミの組織培養の成功にもつながり、Binomica Labsが中心となったカタバミのゲノム解読の原動力になったという。

カタバミの組織培養。コシオバ氏いわく、「カタバミは葉っぱがハート型でかわいいでしょ?」
「読み」のプロジェクト2―デイノコッカス
「デイノコッカス」という放射線耐性がある細菌がいる。これは、放射線だけでなく、高温、低温、乾燥、低圧力、酸といった極限環境にも耐える存在だという。1956年に発見されたデイノコッカス・ラディオデュランス(Deinococcus Radiodurans)が知られているが、デイノコッカス・ラジオフィルス(Deinococcus Radiophlus)、デイノコッカス・プロテオリティカス(Deinococcus Proteolyticus)といった、Deinococcus Radioduransよりもさらに耐性のある細菌も見つかっている。しかし、そのゲノムは解読されていない。
「ちょっと面白そうだし、植物以外もやってみよう。こんな小さな細菌だからすぐにできるだろう」と始めたコシオバ氏らだったが、実際は5カ月もかけてクオリティの高い高分子DNAを抽出することになった。その苦労の甲斐あり、驚くようなリーディングができたという。
近い種である3種のデイノコッカスのシーケンスを見ていくと、それぞれの違いを生み出す要因は「これかもしれない」と見極めできたのだ。このような解き明かしの積み重ねが、進化の謎の解明にもつながっていくのではないか、と氏は考えている。このプロジェクトの詳しい研究成果は、5月に発表される予定だ。


デイノコッカスのリーディングと近種3種(Deinococcus Proteolyticus/Deinococcus Radiophlus/Deinococcus Radiodurans)の相関を比較する円形グラフ(撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])
「書き」のプロジェクト1―スノッズベリー
「青いバラ」といえばサントリーが開発したものが有名だが、コシオバ氏にとって「あれは薄紫にしか見えない」(たいがいの人にとっても同様だ)。青バラにはさほど興味がない氏は、本当に青い色のイチゴを作ってみたいと思った。それは『チャーリーとチョコレート工場』の「スノッズベリー」みたいなもの。「どんな味?」と聞かれても「スノッズベリーみたいな味」としか答えられないようなイチゴだ。
この「書き」のプロジェクトには、熱帯に生息する貝から発見された青色発色タンパク質が使われる。このタンパク質をバクテリアに注入、最適化をはかって37世代進化を続けさせた結果、つい先週に書き換えに成功したという。氏が日本に向けて出発する前日には、このタンパク質「Agroバクテリウム」を組み込んだバクテリアが順調に成育を始めた。あとは植物へのデリバリーシステムの構築となるが、あと6週間くらいで青色のペチュニアが生育するのではないかということだ(ペチュニアは生育が早いのでこのような実験によく使われるらしい)。その次は、青いスノッズベリーの育成に取りかかっていく。
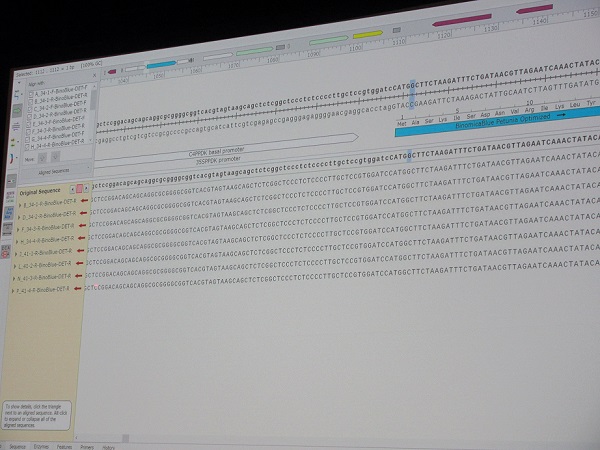
「Agroバクテリウム」組み込みバクテリア「BinoBlue」のゲノム。「上の1行目が期待されるシーケンスで、下の8行がわれわれが到達することができたシーケンスです」
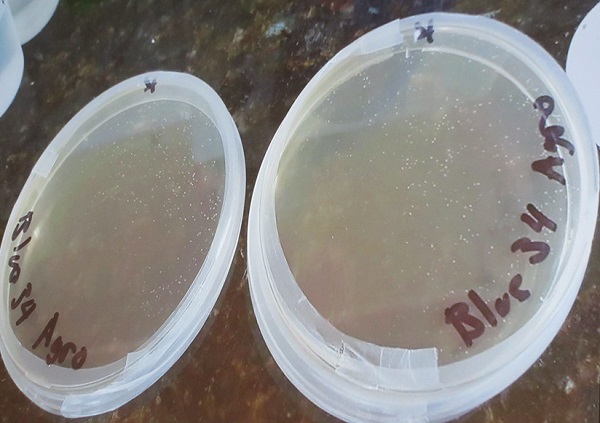
生育し始めた「BinoBlue」。「今回日本に来れてとてもうれしいのですが、すぐにでも飛んで帰ってこれを見守りたいというのも僕の今の気持ち!」
「書き」のプロジェクト2―無限発現システム
最後のプロジェクトについて、コシオバ氏は「さまざまな議論を生んだプロジェクトでした。いろんなことを考えたし、今も考え続けています」と語り出した。
「プロジェクトは、ささやかなきっかけから始まるものです。ある日私たちは、ラボで議論をしていました。そもそも生命体は化学物質から発生している。化学物質がアミノ酸を作り、アミノ酸がタンパク質を構成する。そんなタンパク質は進化とともに次の世代に受け継がれてきた。タンパク質を含むすべての生命体は進化を遂げつつも、淘汰もされてきた。自然界にメソッドはないし、意図的な決定もくだされていないということを前提に考えてはいるのだけれども、さて…『タンパク質発現に偶然はないのか?』『自然界から偶然から取り出せるタンパク質はないのか?』という疑問が生まれたのです」
もちろんこのことを実際に実験して実証していくことは、生命倫理の観点からはグレーゾーンに入ることになる。コシオバ氏は、このプロジェクトを計算上のシミュレーションから開始、そのすべて過程をオープンにして始めることにした。
・ステップ0、1-シミュレーション/機能確認
まずはプログラムを書き、ランダムなDNAからランダムなタンパク質が生み出されるシミュレーションをした。このシステムを使って生み出したシーケンスを解析すると、地球上にあるタンパク質と似ているものはひとつもない。実際に発現させたとして、それが既知の毒性や危険性のあるものと同等の機能を持つかどうか、そこも氏はシミュレーションしてみた。データとして自然界には類似のものがないことが判明したけれど、あくまでも「機能不明のタンパク質」でしかなかった。要するに、わからないものは分析すらできないのである。しかし、“こうして作ったものをバクテリアに注入、タンパク質を発現させることは可能”であり、それがコシオバ氏の強調したい論点なのだ。
・ステップ2、3-切り/貼り
シミュレーション上は類似のタンパク質がないことがわかったけれど、まだデータが足りない。実験を一歩前に進めることにして、カタバミのDNAを切り貼り、架空のタンパク質を「書いて」みることにした。カタバミのDNAを抽出、酵素で塩基の結合をといて「A」「T」「C」「G」とバラバラにする。次は、糊のように機能するDNAリガーゼを用いてランダムに並べ替える。
・ステップ4-クローニング
先ほど並べたATCGの塩基配列とプラスミドの双方に制限酵素の「MlyI」を使い、プラスミドに配列を組み込むと、切り貼りした塩基配列はタンパク質として発現することになる。これを使って、未知のタンパク質を発現する微生物のコロニーができた。「このコロニーは、サイコロを振って出たようなものでしかないんです。けれど、このようなコロニーはできます。しかも、簡単に入手しやすい材料でできるんです」
フリーの構造解析ツールを使って生み出されたタンパク質の構造を見たところ、おおよそ下図のようなものだということがわかった。その他に判明したのは、この構造は「現在地球上にある、いかなるものとも似ていない」ということだった。
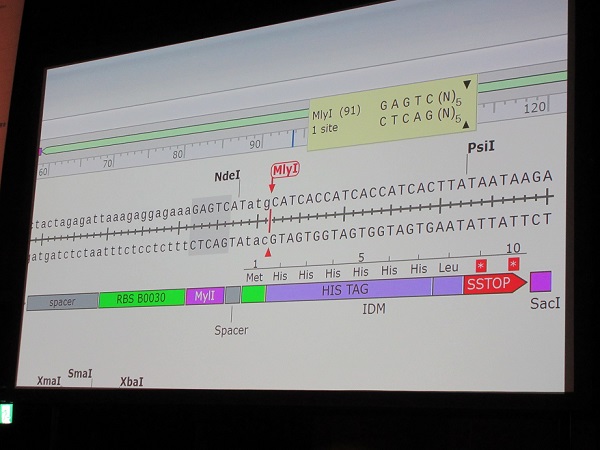
切り/貼りの過程。「図のグレーのところを切って赤いところに貼り付けていくイメージ」であるとのこと


未知のタンパク質によってできたコロニーと、ツールによって描き出された想定の構造
コシオバ氏は強調する。「このプロジェクトは、このようなタンパク質発現がどのような影響をもたらすか、それを知るためのプロジェクトでした」と。コシオバ氏は、機会あるごとに生命倫理の専門家に問いかけもした。「こういうものには無限の可能性があるかもしれない。そこをふまえたうえで、ここから先をどう進めたらよいと思われますか?」とも。様々な答えが返ってきたけれど、通底していた答えは、「結局のところ、やってみなければわからない」だった。コシオバ氏は、思索を続けている。
・可能性として、危険は大きい。農業に非常な被害をもたらすかもしれない、人命がおびやかされるかもしれない。このような実験は、厳密にリスクアセスメント(リスク評価)をしたうえでないとやれはしないのは絶対だ。
・可能性として、科学的な意義も小さくはないはずだ。いまだ謎だらけのタンパク質、その発現や結晶化への探求が可能になるかもしれない。もしかするとこのように新たなタンパク質は何らかの触媒になるかもしれず、例えば新薬開発のターゲットのツールなどになったりはしないか…。
「こうした探求と思索を含めて、Binomica Labsは科学に貢献をし続けていきたいと考えています」とコシオバ氏は、トークを締めくくった。
***
以上のように、今回レポートしたワークショップは、非常に内容が充実したものだった。このほかにも、早稲田大学理工学術院教授でアーティストの岩崎秀雄氏、山口大学工学部応用化学科教授の赤田倫治氏とYCAMバイオ・リサーチのプロジェクトメンバーたちによるトーク、YCAM元チーフエデュケーターの会田大也氏やパフォーマンス集団、contact Gonzoのワークショップ、グループワークもあったのだけれども、記事が長くなりすぎるので割愛させていただく。
すべてのプログラムの終了後に雑談していたところ、「年齢層も職業もばらばらの人たちがいっしょになって未体験のことに取り組む機会なんて、ありそうでない。エキサイティングでした!」という感想を聞いて印象的だった。所属組織や立場を問わず、年齢や専門分野をまたがって「個人化」していくバイオテクノロジーのムーブメント。まさに、ワークショップのテーマである「パーソナル・バイオテクノロジー」を実感した3日間だった。
最後に、この分野における啓蒙と知識の普及になみなみならぬ熱意をもって活動するYCAMのみなさんに敬意を表し、貴重な体験の機会をいただいたことに深く感謝を申し上げます。進みゆくバイオテクノロジーの現実が、より多くの人々の実感とともにありますように。(了)
YCAMのバイオテクノロジー関連イベント(2019年4月~2020年3月)
今年度YCAMは「バイオテクノロジー」をテーマに展覧会やトークイベント、ワークショップなどを展開していく。おもな予定は、以下の通り。
◎「contact Gonzo+YCAMバイオ・リサーチ展」(仮)10月12日(土)~2020年1月19日(日)https://www.ycam.jp/events/2019/contact-gonzo/
アーティスト集団contact GonzoとYCAMバイオ・リサーチが「身体はどこから来て、どこへ行くのか」を出発点に新作インスタレーションを制作。会期中には、ライブ・パフォーマンスやワークショップ、ギャラリーツアーなども開催予定。
◎YCAMバイオ・リサーチ・オープンデイ vol.1~vol.3 10月~2020年1月
◎YCAMオープンラボ2019 11月16日(土)~17日(日)
◎未来の山口の授業 at YCAM 「森のDNA図鑑」 10月13日(日)~11月9日(土)