[編注:レポート#2はこちら]
山口県山口市の「山口情報芸術センター」[通称:YCAM(ワイカム)]は、いわゆるアートセンター活動だけでなく、研究開発にも注力をしている。研究開発チーム「YCAM InterLab」があり、そのチームが企画運営する集中ワークショップが、「YCAM InterLab Camp」だ。今回はその第3弾、「パーソナル・バイオテクノロジー」(2019年3月1日~3日)が開催された。
かつて高度な先端技術だったバイオテクノロジーは、専門外の個人でも関われるほどにすっかり技術やツールがこなれてきている。「DNA解析」や「ゲノム編集」といった専門用語も日常的に耳にするようになってはいる。が、自らサンプルを採取してDNAを抽出してみる、抽出したDNAサンプルからDNAの配列情報を決定する(シーケンシング)といったステップを体験できる機会は、研究室やラボに所属している人でもないかぎり、そうはない。
YCAM InterLabは、2015年に「アグリ・バイオ・キッチン」という講座を経て、年度末に館内バイオラボを設置し、2016年より「YCAMバイオ・リサーチ」プロジェクトをスタートした。2016年から毎年開催している「森のDNA図鑑」というワークショップをはじめ、バイオテクノロジーの実際を一般の人が体験できる機会を設けてきた。特に、子ども向けのバイオ教育プログラムまでを開発、知見を蓄積している点では国内唯一無二だろう。そのYCAM InterLab、とりわけ上述のYCAMバイオ・リサーチを牽引してきたメンバーたちによる「パーソナル・バイオテクノロジー」。ワークショップの内容はいったいどんなものかと少なくない関心が集まり、開催当日はバイオに感度を持つ人たちが全国から山口に参集することになった。
プログラムのスケジュールはおおまかに、1日目「DNAの読み」(サンプル採取からDNA抽出、シーケンシングまでのハンズオン)、2日目「DNAの書き・生命倫理」(バイオインフォマティクスやDNA編集や合成、生命倫理についてのレクチャーとトーク)、3日目「応用可能性の検討」(グループワークとプレゼン)となっている。プログラム内容の魅力とポイントは、おおよそ以下の3点になる。
1.YCAMのこれまでの知見を注入した「読み」のメソッド
「DNAの抽出」と一口に言っても、それは分子の世界。マイクロ、ナノ単位の話である。実験は、マイクロチューブやマイクロピペットを使う繊細な作業の連続だ。それを実験室でもないホールで、ほぼ実験未経験の人たち(今回の参加者は34名)に対して、一斉にレクチャーしつつ進めていく。想像するだに難しいこの挑戦は、野心的ですらある。ただしYCAMは、その運用のために必要な用具や機材、レクチャーのタイミングや順序、スタッフのサポート体制など、経験を積み重ねてきている。YCAMのバイオ関連レクチャーの集大成ともいえる実験の様子については、順を追って後述する。
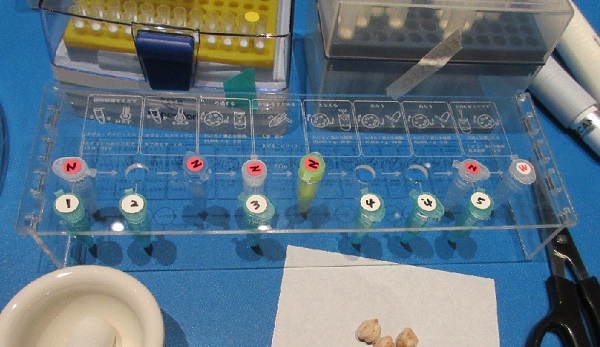
マイクロチューブを立てるラックはYCAM特製。上面のプレートに注目。すごろくのコマを進める感覚で、子どもでも(もちろん不慣れな大人でも!)スムーズに実験を進めていけるよう工夫されている。館内ラボにあるレーザーカッターを使用して作成
2.「ゲノム弁当」なるランチからのサンプル採取
今回サンプルにするのは、植物の細胞組織。ワークショップ期間中のランチに提供される「ゲノム弁当」から採取する。YCAMがベジタブル喫茶「ToyToy」に特注するゲノム弁当は、“ゲノムの解読された生物のみ”を材料にして調理されたお弁当。なぜ、サンプルのゲノム解読/未解読をレクチャーのキーにしているかというと、1日目でたどり着く(予定の)DNA配列データを、2日目に解析するためである。解析のレクチャーでは、データの扱い、必要なプログラムツール、ゲノム情報のデータベースのありか、ゲノムの同定や判断方法の実際を学ぶことになる。そのためには、ゲノム未解読あるいはブラインド状態の細胞組織をサンプルにするより、ゲノム解読ズミで「さっき食べた」ものをサンプルにするほうが、ずっと理解しやすくなる。



これがゲノム弁当。1日目は「ゲノム未解読のスパイス」も含まれるややゆるい弁当。2日目はゲノム解読された食材のみ。3日目は解読年ごとの食材で作られたおかずを、それぞれが取り分けて楽しむブッフェ形式(撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])
3.個人向けDNA解析機材「Bento Lab」のメイカー、個人サイエンティストのトーク
機材には、パーソナルあるいはDIYバイオ向けに開発されて注目されている解析機材「Bento Lab」や、小型シーケンサー「MinION」が多用された。くわえて、Bento Labの開発者であるフィリップ・ボーイング氏を現地に招聘、開発についての貴重な話が聞けた。もう一人、海外からの招聘者には個人サイエンティストのセバスチャン・コシオバ氏がいて、アパートメントの一室をラボにして研究する氏が、小学生の女の子と行う「読み」のプロジェクト、新規のタンパク質発現を試みた「書き」のプロジェクトが紹介された。今まさに進展するバイオテクノロジーの世界の臨場感あふれる二人のトークの内容も、くわしくは後述することにする。

Bento Lab。遠心分離機、PCR、ゲル電気泳動ユニットを備えたオールインワンの遺伝子分析装置となっている。Kickstarterで資金を集めて製品化にこぎ着けた。製品版は今年になって出荷が始まったばかり(撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])
以上の1~3を中心に、今回の「パーソナル・バイオテクノロジー」の様子をお伝えする。
実験の実際――サンプル採取からシーケンシングにたどり着くまで
1日目の午前は、InterLabディレクター伊藤隆之氏より、InterLabの活動や今回ワークショップの目的と日程の説明があり、参加者各人の自己紹介があった。お昼の「ゲノム弁当」を食してからが、いよいよ本番である。本番は、InterLab研究員でYCAMバイオ・リサーチの主軸を担う、津田和俊氏の実験概要説明と各機器の扱い方、諸注意から。と、津田氏がタイムテーブルの目安を話しながら、「事前のテスト演習では、DNAの抽出からシーケンシングに入るまで8時間かかりました」と話すではないか。レクチャーつきで初心者がこの実験を行うと、どうしてもそのくらいはかかる、という。その時点で、時刻はすでに14時をまわっている。単純計算でも終了が22時から早まることはない。しかし、会場からは驚きの声も嘆きの声も聞かれない。そう、人は経験のないことには実感を持てないのだ。初心者たちは「ん? そういうものなの?」と、ただ漠然と思うだけでなのである。
Step1.抽出
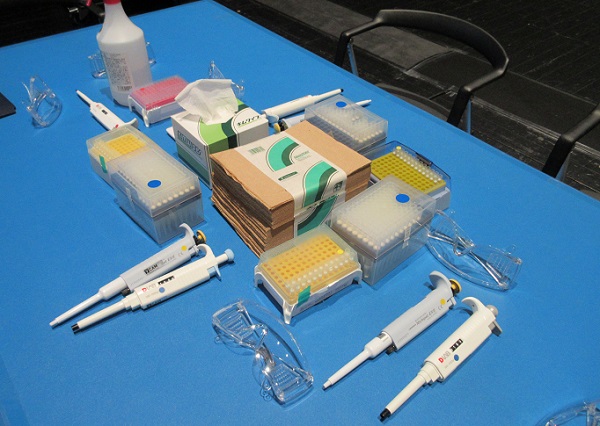
グループごとの各テーブルに整然と配置、準備された実験器具類。ステップに応じて必要になる試薬は、スタッフが都度計量されたものを提供してくれる。各グループは4~5名で、スタッフ1名がつきっきりでサポートしてくれるので心強い

サンプルとなる食材。トマト、ほうれん草、ひよこ豆といった素材14種類、炊き込みごはんの具、コロッケの中身といった料理9種類から選ぶ。すべてゲノム解読ズミ。素材では長い塩基の抽出、料理ものでは混合された塩基の解析を試みる(撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])
・まずは薬包紙の上で、サンプルをできるだけ細断。乳鉢に入れてすりつぶす。その上に液体窒素の代用としてドライアイスを入れて、水分を飛ばしながら粉末状にしていく。

ひよこ豆は細断できず、ハンマーで粉砕して乳鉢に入れることになった。写真はドライアイスとともに粉末にした状態
・粉末状のサンプルをドライアイスと一緒にマイクロチューブに移し、常温あるいはヒートブロックを用いてドライアイスを気化させる(マイクロチューブのフタはしない)。
・400μlのバッファー(界面活性剤など)と4μlのRNase A(リボヌクレアーセ)を加えて、ボルテックスミキサーで撹拌したあと、65度のウォーターバスまたはヒートブロックで10~15分間あたためると、植物組織が溶かされた状態になる。
ウォーターバスに入れる前のサンプルはこんな感じ。必要なサンプル量はほんのちょっぴり、小指の先ほどなのだが、そんなことも体験しないと実感できないものだ
・140μlのバッファー(酢酸など)を加えて、ボルテックスミキサーで撹拌をしたあと、氷の上に5分間静置。これでサンプルは洗われて、DNAと分けられる状態になっている。
・静置後、青色フィルターチューブに移して、Bento Lab最大の8,000xgで遠心する。これでひよこ豆のDNAがろ過された…はず。
・以降、バッファーを加えては遠心、ろ過していく作業が繰り返される。
|DNAをまとめる|分離チューブの底のペレット(かたまり)を崩さないように中味をマイクロチューブに移し、1.5倍量のバッファー(エタノールなど)を加えて混和。
|DNAをとらえる|400μlを黄色フィルターチューブに移して遠心、ろ液を捨てて、元のマイクロチューブに再度セット。
|DNAを洗う|バッファー(エタノールなど)を加えて遠心、ろ液を捨てて元のマイクロチューブに再度セット。
|DNAを洗う2回目|バッファー(エタノールなど)を加えて遠心、新しいマイクロチューブにセット。
|DNAを取り出す|バッファー(EDTA)を黄色フィルターチューブのメンブレンの中心に加え、室温で5分間静置。1分間最高速度で遠心し、マイクロチューブにDNA溶液を回収する。

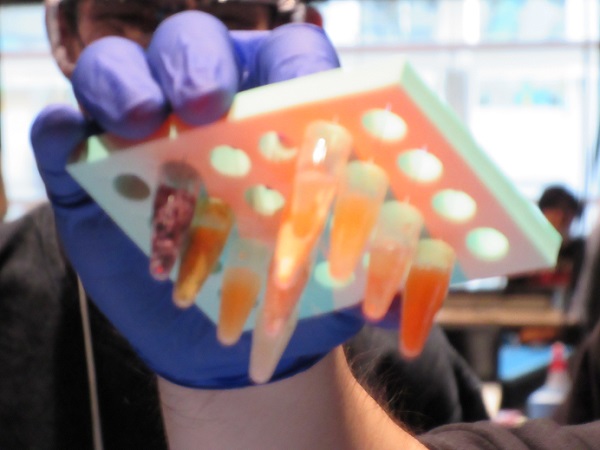
できた溶液。ここにひよこ豆のDNAだけが溶けこんで入っている…はず。肉眼で確認できないためさっぱり実感はないのだが、きっとあるんだろう
Step2.増幅
・PCRチューブの中にDNA溶液を入れ、5種の試薬類(2X PCRマスターミックス、精製水、フォワードプライマー、リバースプライマー、鋳型DNA)を順に注入しながら、ピペッティングまたは軽くタッピングしながら混合していく。
・サーマルサイクラーを設定、98度、64度、72度のサイクルを34回繰り返してPCR(ポリメラーゼ連鎖反応)を実行する。これでDNA断片が増幅されていくはず。

サーマルサイクラーを動かす準備ができたところ。ここから1時間半は機械におまかせ(待ち時間はフィリップ・ボーイング氏のトーク、撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])
Step3.精製
PCR反応産物を精製するため、今回は「AMPureXP」という試薬キットを使う。これは、「SPRI磁性ビーズ」という技術を用いた方法だ。
・PCR反応溶液とAMPureXPを十分に混和し、室温で5分間静置する。これで100塩基以上のPCR反応産物が試薬中の磁性ビーズに結合するという。

PCR反応産物が磁性ビーズと結合した状態。このマグネットスタンドもレーザーカッターで作られたYCAM特製。向こう側には結合物を吸い寄せるための磁石がペタリと貼られていて、側面のYCAMロゴもいいね!(撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])

PCRチューブをマグネット側に設置して2分間静置すると、磁性ビーズが引き寄せられて溶液が透明になる。この茶色いカスみたいなのが、われわれが求めてきたとても大事なもの!(撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])
・溶液を捨て、磁性ビーズを70パーセントエタノールで2回洗浄する。
・PCR反応産物を溶出するために溶出バッファー(精製水)をチューブに添加、ピペッティングしてよく混ぜる。これを再びマグネットスタンドに立てると、今度は結合がはずれた磁性ビーズだけが磁石に吸い寄せられる。
・増幅されたDNAの入った溶液を取り出す。今度の溶液を捨ててはいけない! 絶対に!
Step4.定量
ここからようやく、DNAの姿が見えてくる(ただしデータで)。NanoDrop OneC分光光度計を使ってサンプル溶液を定量分析することにより、サンプル内のDNA、RNA、タンパク質の量がわかるのだ。

ここまで数時間かけて実験してきた結果の溶液、1~2マイクロリットルを台座に滴下すると、溶液内に抽出されたDNAがちゃんと含まれているかどうかが、わずか数秒でわかる!(撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])
Step5.シーケンシング
通常、ラボや研究室の「読み」の作業では、前の定量までのステップをこなし、その後のシーケンシングは外部のシーケンシングサービスに依頼することが多い。最近、サービスは1件350円前後までコストダウンしているというから、それも納得だ。今回のワークショップは、シーケンシングも自前で行う。それも、ナノポア(ナノスケールの穴)にDNA鎖を通すという技術を活用したパーソナルな小型シーケンサー「MinION(ミナイオン)」を使ってやってみることになる。

このMinION、ベーシックなスターターキットでもまだ高価だ(ン万円)。繰り返し使えるのはガワのホルダー部分だけで、肝心のフローセル部分は1回しか使用できない(96サンプルを1回で読む方法もあるそうだ)。またつい最近、使い捨てフローセル(商品名Flongle)がひとつ90ドル程度で発売された。改良と低コスト化は、日進月歩で進んでいる!、撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])

小さな精密機器であるMinIONの分析精度を上げるには、マイクロピペットを使いこなして流路を確保するなど、いくつかのコツがいる。「高いんだから…失敗できないよ!」のスルドい視線を四方からあびつつ作業する彼女だって、ついさっき初めてマイクロピペットに触ったばかりなのね(撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])
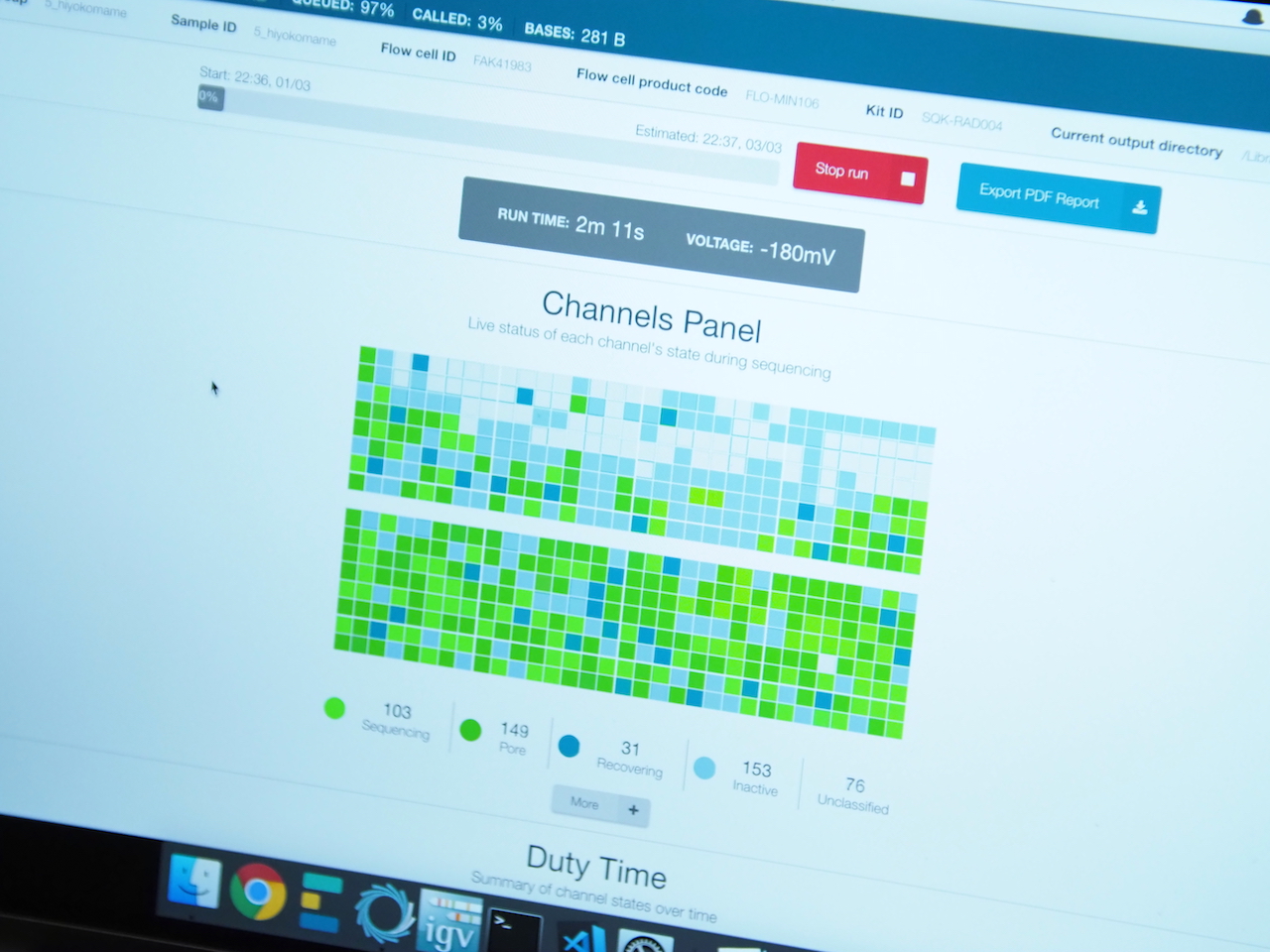
DNA溶液をフローセルに滴下、ポートのカバーを閉め、プライミングポートを閉め、MinIONのフタを閉め…と順番におこなう。PCのビューワーの実験条件などの設定をすましてしばらくすると、MinIONで塩基データが読まれている様子が出てきた!(撮影:田邊アツシ、写真提供:山口情報芸術センター[YCAM])
MinIONの稼働前後には、各グループのテーブルから「おおぉー!!」という歓声、拍手がわき起こった。体験していなかったことを実際に体験すると、人は「なるほど、こうだったのか!」を実感して感動するのだ。
22時はとっくにまわっていて、津田氏の宣言通りの「8時間以上」の実験プログラムは無事に終了。MinIONとPCが一晩稼働し続けたあとの結果がどうなっているか、そのデータをどう解析するかは、2日目以降のお楽しみとなった。
スタッフに聞いたところによると、子ども対象のDNA抽出のワークショップでは、子どもたちは初めてでも上手にマイクロピペットを操りながら案外スイスイと実験をこなしていくそうだ。理由は、老眼とは無縁な「よく見える目がある」から! そういえば、DNAの二重らせん構造のX線撮影に成功したロザリンド・フランクリンの伝記には、「とても手先が器用な人だった」という話もあったなぁ。実感や感動や感慨やのあれこれを味わいつつ、チューブ内の1マイクロリットルの液体(ほんのちょっぴり)と格闘した1日目だった。
と、まずは1日目の実験の様子をお届けしました。2日目以降の内容は、この記事の続編として、データベースの専門家片山俊明氏によるゲノムデータの解析とテクニックの紹介、フィリップ・ボーイング氏とセバスチャン・コシオバ氏の刺激的なトークを中心にお届けする予定です。(つづく)